Allgemeines zur DNA
Die Nukleinsäurenbasen sind die Grundlage der DNA. Man unterscheidet 4 verschiedene Basen für die DNA, respektive die RNA. Die Basen paaren sich nach einem ganz bestimmten Schema: Das Purin
Adenin paart mit dem Pyrimidin Thymin über 2 Wasserstoffbrücken im „Innern“ der Doppelhelix während Guanin mit Cytosin eine dreifache „Bindung“ mittels Wasserstoffbrücken
eingeht. Die Guanin-Cyotosin Bindung ist aus diesem Grund stabiler. Dieses Paarungsschema, benannt nach Watson + Crick, ist Grundlage für die Ausbildung der Doppelhelix der DNA. Die einzelnen Stränge
der DNA sind antiparallel angeordnet. Jeder Strang weist eine Polarität auf in Form eines 5’ und eines 3’ Endes. Diese Nomenklatur beruht auf der Verknüpfung der Desoxyribosen
und der Phosphate untereinander, die das negativ geladene Rückgrat der Doppelstränge bilden. Antiparallel bedeutet hier also, dass der eine Strang von 5’ nach 3’ läuft, während
der andere mit 3’ beginnt und mit 5’ endet. Interessant zu wissen ist, dass Alterung mit der Polarität der Stränge zusammenhängt! Mehr dazu folgt jedoch im nächsten Jahr.
Verschiedene Formen der DNA
Man unterscheidet mehrere Formen der DNA:
- B-Form: 10,4 Basenpaare pro Windung
Rechts drehend, d.h. im Uhrzeigersinn (wenn man in die 5’-> 3’ Richtung schaut)
Basenpaarung nach Watson + Crick ( 5’- 3’ Anordnung)
3,4 Angström Abstand zwischen zwei Basen ( =p)
99% der menschlichen DNA
- Z-Form: 12 Basenpaare pro Windung
links drehend
Funktion noch unklar
In einem Strang kommen repetitiv je ein Purin und ein Pyrimidin vor
Beispiele von Strängen die eine Z-DNA bilden können: (C G)n oder ( A C)n
- A-Form: 11 Basenpaare pro Windung
Rechts drehend
Um die einzelnen Stränge zu analysieren muss die Doppelhelix getrennt oder denaturiert wer-den. Dies kann durch erhöhen der Temperatur erreicht werden. Man misst die „Schmelz-temperatur“ der
DNA. Die Tm (temperature of melting) ist definiert als diejenige Temperatur bei der 50% der DNA als Einzelstränge vorliegt. Gemessen wird die Tm mit Hilfe der Licht-absorption bei der Wellenlänge
260 nm. Doppelsträngige DNA (dsDNA) absorbiert weniger Licht als Einzelsträngige (ssDNA). Dieser Effekt wird Hyperchromizität genannt.
Die Tm ist abhängig :
- von der Art der DNA
- vom G-C Gehalt
- von der Länge (nur bei weniger als 100 Basenpaaren)
- von der Beigabe von Na+, welches an die negativen Phosphate bindet und die Abstossung unter den Phosphat-Gruppen senkt
Wer die Tm ganz genau berechnen will, für den folgt hier die Formel für eine Doppelstrang- DNA (ds DNA) mit > 100 bp:
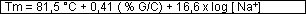
Für alle anderen Medis gilt zu wissen, dass normale Tm jeweils um 80°C liegen. Unter optimalen Bedingungen (optimal annealing temperature = ca. Tm – 25 °C) finden sich entartete Einzelstränge wieder zur Doppelhelix!
Die DNA-Tripelhelix
Neben der Doppelhelix findet man auch Tripelhelices, bei der 3 Stränge gepaart sind. Tripelhelices werden häufig künstlich für medizinische Zwecke hergestellt. Für die Ausbildung
einer Tripelhelix ist wichtig, dass in einem Strang nur Purine und im anderen nur Pyrimidine vorkommen. Bei dieser Anordnung der Stränge kann sich ein weiteres Oligonucleotid in der Major Groove,
der grösseren Rille, der Doppelhelix einlagern. Bei der schraubenförmig gedrehten Doppelhelix unterscheidet man zwischen einer grossen Rille und einer kleinen Rille. Die Rillen liegen zwischen
den Doppelsträngen und nicht zwischen den einzelnen Strängen! Die Oligonucleotide können aktivierend oder hemmend auf die „DNA“ wirken. Die Wirkung gewisser Medikamente
beruht auf dieser Eigenschaft.
Da in einer Tripelhelix drei negative geladene Phosphatketten der Rückgrate zusammen-kommen, stossen sie sich gegenseitig ab und die Tripelhelix ist nicht sehr stabil. Um die Abstossung zu vermeiden,
wird ein künstliches Rückgrat aus Proteinen verwendet, welches die üblichen Basen trägt. Das künstliche Trägerskelett wird PNA (peptid nucleic acid) genannt. Diese künstlichen
Tripelhelices sind sehr stabil, da keine Abstossung aufgrund der Phosphoribosen vorkommt und der Körper sie nicht enzymatisch abbauen kann!
Neben der Tripelhelix können auch Helices mit vier oder fünf Strängen synthetisiert werden.
Elektrophorese von DNA
Da DNA normalerweise viel zu lang ist um sie am Stück zu sequenzieren und analysieren, muss sie erst fragmentiert werden. Die Fragmente werden nachher bei pH 8, bei pH 8 ist die DNA negativ
geladen, auf ein Agarosegel aufgetragen. Die Fragmente wandern zur positiven Anode. Das Agarosegel bremst die Fragmente proportional zur ihrer Grösse. Je kleiner die Fragmente sind umso grösser
ist die Migrationsgeschwindigkeit.
Um die DNA Fragmente sichtbar zu machen, kann fluoreszierendes Ethidiumbromid in das Agarosegel beigefügt werden. Das (mutagene) Ethidiumbromid interagiert hydrophob mit den Basenpaaren und
dabei erhöht sich die Fluoreszenz stark. Unter UV Licht ist somit die DNA aufgrund der höheren Fluoreszenz gut sichtbar. Um die Anzahl Basenpaare zu bestimmen werden Marker mit bekannter
Länge parallel in Gel laufen gelassen. Sie ergeben sehr genaue Anhaltspunkte im Agarosegel.
Eine Elektrophorese menschlicher DNA ist nicht möglich, da sie zu komplex ist und zu viele Fragmente bei der Spaltung mit Hilfe von Restriktionsenzymen ( siehe unter Plasmid DNA) entstehen. Auf
einem Agarosegel „hinterlässt“ menschliche DNA nur einen Streifen („smear“) von oben bis unten. Eine Bestimmung des Molekulargewichtes der einzelnen Fragmente ist somit
nicht möglich. Vereinzelt findet man dickere Querbalken im grossen Streifen. Dabei handelt es sich um repetitive Sequenzen im menschlichen Erbgut, sogenannte „tandem repeats“, die
alle die gleiche „Grösse“ haben.
Plasmid DNA
Ein Plasmid besteht aus einer kreisförmigen Doppelstrang-DNA, die in sich verdreht sein kann. Ein Plasmid verfügt über 1000-100000 Basenpaare, die Grösse ist also sehr variabel.
Auf dem Doppelstrang findet man eine Sequenz, die Ori (origin of replication) genannt wird und die Anfangssequenz für die Replikation darstellt, wie der Name schon sagt. Natürliche Plasmide
stammen v.a. aus Bakterien. Auf Plasmiden befinden sich oft Antibiotika Resistenzgene, die leicht von einem Bakterium auf einen anderen übertragen werden können.
Restriktionsenzyme oder Restriktionsendonucleasen: besitzen die Fähigkeit, doppelsträngige DNA nach ganz spezifischen Basensequenzen, sog. Palindromenen, von mindestens vier Basenpaaren
zu schneiden. Entweder entstehen dabei überlappende Enden („sticky ends“) oder eine glatte, gerade Trennung („blunt ends“). Die „Immunabwehr“ von Bakterien
beruht auf diesem System. Die Restriktionsenzyme wurden bei der Untersuchung dieser „Immunabwehr“ der Bakterien entdeckt.
Ein Beispiel für ein Restriktionsenzym ist Eco RI aus dem Bakterium E. coli. Es erkennt die Basensequenz GAATTC und schneidet an dieser Stelle. Da die Stränge der DNA komple-mentär
sind, werden beide Stränge versetzt geschnitten Þ sticky ends (die später wieder „zu-sammenkleben“ können). Je häufiger diese Sequenz vorkommt, desto mehr Fragmente
entstehen.
Mit Hilfe von Ligasen, d.h. bestimmten Enzymen, können Fragmente bei tiefer Temperatur unter Energieverbrauch wieder zusammengefügt werden. Ligasen benötigen Energie (ATP oder NAD+
je nach Spezies)
Wird mit einem Plasmid eine Elektrophorese gemacht, stellt man fest, dass das ungeschnit-tene Plasmid ³ zwei verschiedene Wanderungsgeschwindigkeiten im Agarosegel aufweist. Die Masse des Plasmid
bleibt immer gleich, doch es ändert seine Anordnung im Raum (Þ verschiedene Topomere) und erfährt so mehr oder weniger Reibung im Gel. Das Plasmid kann sich um sich selber drehen und
so eine schraubenförmige Gestalt annehmen. Das Plas-mid ist super-coiled. Ist das Plasmid nicht verdreht spricht man vom relaxed Zustand. Das super-coiled Plasmid wandert im Gel am schnellsten.
Die DNA der Wirbeltiere ist entlang einer Matrix aus Proteinen in Schleifen von 500- 1000 Kilobasenpaaren angeordnet. Durch diese Fixierung entstehen (beinahe) zirkuläre DNA- Doppelstränge,
die wie Plasmide super-coiled sein können. Man unterscheidet zwischen positive und negative super-coil. Links herum (left handed) verdreht entspricht positiv super-coiled und rechts herum ( right
handed) verdreht dementsprechend negativ super-coiled.
Was ist aber der Unterschied zwischen positiv und negativ und wo spielt das eine Rolle? Während der Replikation und Transkription wird der Doppelstrang geöffnet. Dabei entsteht im Rest
der zirkulären DNA eine positiv super-coiled DNA, was einer starken Verdrehung entspricht. Wird die Verdrehung zu stark, kann der Doppelstrang nicht mehr geöffnet werden und die Replikation
resp. Transkription wird behindert. Liegt die zirkuläre DNA nun vor der Replikation / Transkription im negativ super-coiled Zustand vor, wird zuerst der relaxed Zustand erreicht und erst danach
beginnt die Umwandlung in den positiv super-coiled Zustand. Die negativ super-coiled Form stellt somit eine Energiereserve dar, die allerdings unter Energieverbrauch von Topoisomerasen (Enzymen) hergestellt
wurde. Eine negative super-coiled DNA lässt sich somit für die Replikation / Transkription leichter öffnen.
Wie ist nun die menschliche DNA auf die Histone aufgewickelt? Die DNA ist left handed um die Histone gewickelt, damit sie bei Entfernen der Histone für die Replikation / Transkription eine right
handed oder negativ super-coiled Form einnimmt!
Southern Blot
Die Methode des Blottens dient zum finden eines bestimmten Fragmentes einer DNA oder ei-ner bestimmten Sequenz in einem Fragment. Das Suchen der Sequenz geschieht mit Hilfe ein-er Sonde, d.h. einer
markierten und bekannten single stranded DNA oder RNA. Diese Sonde sucht ihren Partnerstrang unter den fixierten und getrennten DNA Strängen nach den Watson-Crick Kriterien der Basenpaarung.
Da das Agarosegel dafür zu dick ist, werden die DNA Stränge nach der Denaturierung mit NaOH (Þ single strand Bahnen) auf ein Nitrocelluloseblatt übertragen („blotten“).
Dies er-folgt durch Diffusion oder im elektrischen Feld. Diese single strands werden nun mit den markierten Sonden hybridisiert. Hybridisieren bedeutet renaturieren nicht mit dem ursprüng-lichen
Partner. Die Sonde findet den komplementären Strang. Durch Kontrolle der Tem-peratur kann eine stabile Bindung gewährleistet werden. Nach waschen der Folie kann die ge-suchte DNA auf der
Folie detektiert und somit auf dem Gel rückwirkend lokalisiert werden. Je nach verwendetem Untersuchungsmaterial und Sonde, werden die Blots anders benannt, doch die Methode ist jeweils die gleiche!

Die letzten beiden Blots sind allerdings sehr selten!
Chemische Sequenzierung der DNA
Die DNA Fragmente werde z.B. am Ende markiert. In der Regel ist dies das 5’- Ende. Die Markierung gibt somit die Ablesungsrichtung des Fragmentes bekannt.

Das Fragment wird nun mittels eines Enzyms geschnitten und nachher erfolgt eine Trennung durch Elektrophorese. Dabei können nur die markierten Schnittfragmente detektiert werden!

Nun wird eine chemische Substanz beigefügt, die spezifisch mit einer der vier Basen reagiert, beispielsweise mit der Base Guanin (G). Die Reaktion führt zur Spaltung des Stranges nach dem
G. Die Bedingungen werden so gewählt, dass pro Strang nur wenige Gs, ausgewählt nach dem Zufallsprinzip, reagieren.
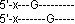
Es entstehen verschieden lange Fragmente, die alle mit einem Guanin enden. Die Bedingungen sind so gewählt, dass alle möglichen mit einem G endenden Fragmente entstehen!
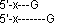
Das Ganze wird nun mit allen vier Basen wiederholt und die Fragmente werden in vier Bahnen auf ein Elektrophorese Gel aufgetragen. Die Fragmente folgen sich nach ihrem Länge und erlauben so
eine Sequenzierung der einzelnen Basen. Da chemische Agenzien oft nach zwei und nicht nur nach einer Base trennen, kann eine Elektrophorese wie folgt aussehen:

Enzymatische Sequenzierung (= Kettenabbruch Methode)
Vorgehen in einzelnen Schritten:
- man nehme eine ss-DNA
- Hybridisierung mit einem bekannten Oligopeptid (= Primer), welches den Start für die DNA- Polymerase darstellt
- Polymerase kopiert den Strang komplementär (= Primer-Elongation)
- Herbeiführen eines künstlichen Abbruches durch Beifügen eines „gefärbten“, d.h. eine fluorsezierende
Gruppe tragenden Didesoxynucleotides (ddNTP), an welches kein weiteres Nucleotid mehr angehängt werden kann
- Durch Beigabe kleiner Mengen ddATP wird erreicht, dass man alle möglichen Frag-mente,
die mit ddA enden, erhält.
- Ein Abbruch wird ebenfalls mit ddGTP, ddCTP und ddTTP herbeigeführt
- Alle Fragmente werden auf einer Bahn in der Elektrophorese laufen gelassen, da die Endbasen der Didesoxynucleotide unterschiedlich „gefärbt“ sind Þ ablesen
der letzten Basen = Sequenz der DNA, da die Fragmente der Grösse nach auf einer einzigen Bahn im Gel aufgespalten werden
Schritte 1-4 sind eine spezielle Anwendung der PCR (polymerase-chain-reacttion)Technik!
|
